Выделение и идентификация нетуберкулезных микобактерий у пациентов фтизиатрических учреждений 03. 00. 07 Микробиология
| Вид материала | Автореферат |
- Бактериологическая диагностика туберкулеза внелегочных локализаций с исследованием, 271.98kb.
- Идентификация и внутривидовое типирование возбудителей сапа и мелиоидоза 03. 00., 750.11kb.
- Омелотерапия в онкологии с точки зрения доказательной медицины, 135.77kb.
- Цивилизации древнего востока, 288.82kb.
- Примерная программа дисциплины микробиология, вирусология, микробиология полости рта, 447.35kb.
- «Микробиология», 161.59kb.
- Контрольная работа по дисциплине Микробиология По теме Общая и специальная микробиология, 208.97kb.
- Резолюция второго всероссийского конгресса пациентов город Самара 03 ноября 2011, 206.55kb.
- Вопросы к экзамену по дисциплине «Микробиология, физиология питания, санитария», 45.83kb.
- Рабочей программы учебной дисциплины (модуля) микробиология, 48.16kb.
* В 14 пробах – с MAC и в 6 – с M. fortuitum
** В 14 пробах – с M. tuberculosis и в 2 – с M. fortuitum
*** В 2 пробах – с MAC и в 6 – с M. tuberculosis
**** Прочие (M. gastri, M. simiae – 2, M. szulgai, M. terrae, M. malmoensae, M. smegmatis)
Самым распространенным видом оказались M. avium complex, что согласуется с известным (по данным зарубежных авторов) значением МАС в патологии человека. Вторыми по частоте выделения были M. fortuitum (однако, как одно-, так и неоднократное выделение быстрорастущих микобактерий, требует тщательного изучения наличия и особенностей симптомов заболевания для оценки их этиологической значимости). Затем следовали M. kansasii и M. xenopi. У двоих больных M. xenopi были обнаружены в операционном материале (содержимое туберкулемы, каверны), что даже при однократном обнаружении может служить подтверждением диагноза микобактериоза.
232 культуры микобактерий в результате комплексного микробиологического исследования были отнесены к M. tuberculosis. В данном разделе работы речь идет о тех культурах, которые первоначально вызвали трудности при дифференциации от НТМ. В первую очередь, это культуры, с не типичной для M. tuberculosis морфологией колоний, выделенные на среде Л-Й от больных, длительно получавших противотуберкулезную терапию. В жидкой питательной среде – контаминированые посторонней микрофлорой или смешанные культуры микобактерий (M. tuberculosis + M. fortuitum, M. tuberculosis + МАС). При микроскопии мазков из таких культур не наблюдалось характерного расположения клеток в виде «кос» – проявление корд-фактора при росте на жидких питательных средах и морфология клеток M. tuberculosis была измененной.
При анализе частоты и видового состава НТМ в зависимости от вида исследуемого материала, установлено, что большая часть НТМ была выделена из мокроты (что вполне понятно, поскольку этот источник является наиболее доступным), как «спонтанной» (без каких либо «местных» воздействий), так и после ингаляции. Реже НТМ обнаруживали в материале бронхоальвеолярного лаважа и бронхиальных смывов, а также в других образцах (плевральный экссудат, материал биопсии и др.). При этом не было существенных (статистически достоверных) различий в соотношении частоты выявления разных видов НТМ в зависимости от источника исследуемого клинического материала.
Проанализирована также частота выделения НТМ в зависимости от диагноза, с которым материал поступил на исследование из противотуберкулезных диспансеров. Выявлены некоторые особенности в распределении видов НТМ по группам с диагнозом: туберкулез, нетуберкулезные заболевания легких, «диагностический поиск». Так, M. kansasii несколько чаще обнаруживали в «диагностическом» материале, чем у больных с диагнозом «туберкулез», а МАС и M. xenopi у больных с клиническим диагнозом «туберкулез».
Изучена частота неоднократного выделения от больных одного и того же вида НТМ (рис. 2). Эти результаты важны, так как неоднократное обнаружение НТМ с большой вероятностью указывает на его этиологическую роль в развитии микобактериоза у данного больного.
Всего НТМ повторно обнаружены у 114 больных; в 47 случаях (41,2%, что достоверно больше, чем другие виды микобактерий, р < 0,05) это были МАС, в 24 (21,1%) – M. kansasii, в 25 (21,9%) – M. fortuitum, в 12 (10,5) – M. xenopi, в 4 (3,5%) – M. flavescens и по 1 случаю (0,9%) – M. gordonae и M. smegmatis.
Двукратно выделяли один и тот же вид НТМ от 73 больных (64,0%), трехкратно – от 29 (25,4%) и более четырех раз – от 12 (10,5%) больных.
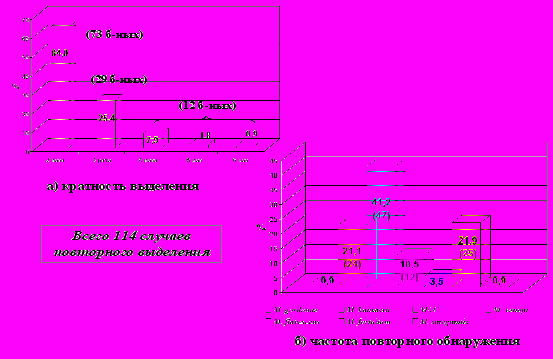
Рис. 2. Частота повторного выделения нетуберкулезных микобактерий
Здесь имеет смысл особо обратить внимание на случаи трехкратного и более обнаружения НТМ у одного и того же больного. По критериям Американского торакального общества (Wallace R.et al., 1997) это является (при наличии клинико-рентгенологической симптоматики) важным диагностическим критерием микобактериоза. Всего трехкратно и более НТМ были выделены от 41 больного – 36,0% всех случаев их неоднократного обнаружения (в т.ч. MAC – от 20, M. kansasii – от 8, M. fortuitum – от 8, M. xenopi – от 4, M. flavescens – от одного больного).
2. Применение метода высокоэффективной жидкостной хроматографии (ВЭЖХ) для определения вида НТМ
В этом разделе работы сопоставлены результаты определения вида микобактерий микробиологическими методами и ВЭЖХ. Содержание миколовых кислот, анализируемых методом ВЭЖХ, и их соотношение в бактериальной клетке является устойчивым фенотипическим признаком, характерным для каждого вида микобактерий, что позволяет получать достоверные, воспроизводимые результаты. Высокая чувствительность метода за счет применения флуоресцентного детектора дает возможность анализировать культуры, выделенные в жидких питательных средах, даже с небольшим содержанием бактериальных клеток, что значительно сокращает сроки идентификации.
На рисунке 3 в качестве примера представлены хроматограммы, полученные при анализе миколовых кислот музейного штамма M. avium ATCC35712 и штамма, выделенного от больного.
Совпадение результатов, полученных с помощью ВЭЖХ и бактериологических методов, имело место в 94,4% случаев; в том числе для медленнорастущих НТМ – в 93,4%, быстрорастущих – в 87,5% (в целом для НТМ – в 92,4%), а M. tuberculosis complex – в 99,0% (табл.8.). При этом следует подчеркнуть, что идентификация вида НТМ культуральными и биохимическими методами занимает до трех недель, тогда как применение ВЭЖХ сокращает это время до 24 часов (статистически достоверные различия результатов идентификации микобактерий микробиологическими методами и ВЭЖХ отсутствуют).
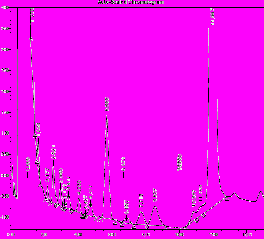
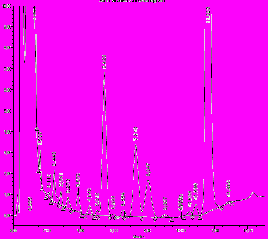
-
а
б
Рис. 3. Анализ миколовых кислот M. avium с помощью метода ВЭЖХ: а – M. avium АТСС 35712 (из музея МНПЦБТ); б – культура № 4-M. avium, выделенная от больного
Таблица 8.
Сравнительные данные идентификации микобактерий
микробиологическими методами и ВЭЖХ *
| Группа микобактерий | Вид | Методы | % совпадений | |
| микробиологические | высокоэффективная жидкостная хроматография | |||
| M. tuberculosis complex | M. tuberculosis | 98 | 97 | 99,0 |
| M. bovis | 2 | - | ||
| M. bovis BCG | - | 2 | ||
| Медленнорастущие нетуберкулезные микобактерии | МАС | 94 | 89 | 94,7 |
| M. kansasii | 30 | 28 | 93,3 | |
| M. xenopi | 37 | 35 | 94,6 | |
| M. gordonae | 15 | 13 | 86,7 | |
| M. scrofulaceum | 3 | 2 | 66,7 | |
| M. szulgai | 1 | 1 | | |
| M. marinum | 1 | 1 | | |
| M. gastri | 1 | 1 | | |
| M. terrae complex | 1 | 1 | | |
| Всего | 183 | 171 | 93,4 | |
| Быстрорастущие нетуберкулезные микобактерии | M. fortuitum | 30 | 28 | 93,3 |
| M. chelonae | 5 | 5 | 100,0 | |
| M. flavescens | 4 | 1 | 25,0 | |
| M. smegmatis | 1 | 1 | | |
| Всего | 40 | 35 | 87,5 | |
| Всего НТМ | 223 | 206 | 92,4 | |
| ИТОГО | 323 | 305 | 94,4 | |
* В таблице за 100% взяты результаты микробиологического исследования, с которыми сопоставлены данные ВЭЖХ (в таблице не учитывали, что в ряде случаев окончательная идентификация была основана на результатах использования комплекса методов, включая ВЭЖХ и молекулярно-генетические исследования)
Проведено также сопоставление результатов идентификации методом ВЭЖХ культур микобактерий, выделенных в жидкой и на плотных питательных средах. Всего были проанализированы 243 культуры (сопоставляли данные идентификации только наиболее часто выделяемых культур). Установлено, что в целом совпадение результатов, полученных двумя методами при идентификации культур, выделенных в жидкой питательной среде, имело место в 93,4%, а на плотных – в 93,0% случаев (табл.9), в одном случае M. fortuitum, выделенные на среде Л-Й, методом ВЭЖХ идентифицировать не удалось.
Таблица 9.
Результаты идентификации микобактерий, выделенных на плотной (Л-Й) и в жидкой (7Н9) питательных средах, с помощью ВЭЖХ
| Результаты микробиологических исследований (число выделенных культур) | Высокоэффективная жидкостная хроматография, культуры со среды: | |||
| Левенштейна-Йенсена | Миддлбрука 7Н9 | |||
| абс. | % совпадений | абс. | % совпадений | |
| MAC (78) | 74 | 94,9 | 74 | 94,9 |
| M. fortuitum (65) | 60 | 92,3 | 61 | 93,8 |
| M. xenopi (38) | 35 | 92,1 | 35 | 92,1 |
| M. kansassii (32) | 30 | 93,8 | 30 | 93,8 |
| M. chelonae (12) | 11 | 91,7 | 11 | 91,7 |
| M. gordonae (11) | 10 | 90,9 | 10 | 90,9 |
| M. flavescens (7) | 6 | 85,7 | 6 | 85,7 |
| ВСЕГО (243) | 226 | 93,0 | 227 | 93,4 |
Таким образом, независимо от того проводили анализ с помощью ВЭЖХ изолятов, выделенных в жидкой или на плотной питательных средах, получены практически идентичные данные, однако, использование культур с жидкой среды предпочтительнее из-за сокращения сроков получения результатов.
3. Молекулярно-генетические исследования для идентификации НТМ
Молекулярно-генетические методы в настоящее время широко используются для видовой идентификации микроорганизмов. После того, как был полностью расшифрован геном ряда микобактерий, произошел существенный прогресс в разработке методов их идентификации и соответствующих диагностических тестов. Достаточно сложной задачей является поиск геномной мишени, при молекулярно-генетическом анализе которой будет возможна идентификация вида НТМ (выбор мишени осложняется большим сродством геномов различных представителей рода Mycobacterium). В настоящее время, для этой цели используют инсерционные последовательности (IS900, IS901, IS1245, IS1561, IS2404), структурные гены (rpoB, gyrB, hsp65, secA1, recA, sodA, ген 16S rRNA, область ITS гена 16S rRNA) микобактериальной ДНК.
На основе изучения микобактериального генома и исследований возможных генетических мишеней, приемлемых для видовой идентификации микобактерий, разработаны различные методы молекулярно-генетического определения вида микобактерий: полимеразная цепная реакция (ПЦР), ПЦР в сочетании с гибридизационным анализом, гибридизация in situ, ПЦР в сочетании с рестрикционным анализом (полиморфизм длин рестрикционных фрагментов), секвенирование. Чаще всего с этой целью используют ПЦР в сочетании с гибридизационным или рестрикционным анализом.
В настоящем исследовании для определения вида НТМ были использованы два молекулярно-генетических метода – биологические микрочипы, разработанные совместно с сотрудниками Института молекулярной биологии им. В.А. Энгельгардта РАН и вариант определения полиморфизма длин рестрикционных фрагментов гена hsp65.
3.1. Разработка и испытание биологического микрочипа (биочипа) для идентификации НТМ
Одним из интенсивно развивающихся направлений биотехнологии нуклеиновых кислот в последнее время является использование микроматриц ДНК для анализа нуклеотидных последовательностей. В этой группе методов на небольшой по размеру поверхности стекла или другого твердого носителя иммобилизуют в виде правильно расположенных микропятен небольшие фрагменты ДНК с известной последовательностью нуклеотидов, которые далее используют для гибридизации с анализируемыми образцами нуклеиновых кислот. При совпадении первичной структуры ДНК микропятна и анализируемого образца на поверхности стекла образуются правильные ДНК-ДНК гибриды, которые обнаруживают по появлению сигналов в данных участках микроматрицы, например, в виде микроскопической флуоресцирующей точки. Твердые носители с нанесенными на них микроматрицами ДНК получили название микрочипов ДНК.
В настоящее время в Российской Федерации лицензированы две тест-системы с использованием биологических микрочипов, которые применяются во фтизиатрической практике. Это тест-система «ТБ-БИОЧИП», позволяющая одновременно выявлять M. tuberculosis в клиническом материале и определять их устойчивость к рифампицину и изониазиду, и тест-система «ТБ-БИОЧИП-2» – для определения лекарственной устойчивости M. tuberculosis к фторхинолонам (обе тест-системы разработаны в Институте молекулярной биологии им. В.А.Энгельгардта РАН, совместно с МНПЦБТ).
Идея создания биологических микрочипов для определения видов НТМ возникла в связи с нарастающей актуальностью проблемы их идентификации и ускоренной лабораторной диагностики микобактериозов.
Данный метод основан на анализе видоспецифического участка гена gyrB, размером 371 п.н. и сегодня позволяет идентифицировать девять видов НТМ (M. avium, M. intracellulare, M. sсrofulaceum, M. kansasii, M. gordonae, M. xenopi, M. marinum, M. fortuitum, M. chelonae) и дифференцировать их от M. tuberculosis complex (M. tuberculosis, M. bovis, M. bovis BCG). Разработку метода проводили поэтапно, с последовательным увеличением количества иммобилизированных в заполненных гелем лунках биочипа специфических для разных видов микобактерий олигонуклеотидов. ДНК-ДНК гибридизация происходит в трехмерном измерении, что является отличительной особенностью данного вида биочипов от используемых за рубежом.
Ген gyrB ответственен за синтез β-субъединицы фермента ДНК-гиразы (топоизомеразы II типа), которая индуцирует образование отрицательных сверхвитков в релаксированной кольцевой молекуле ДНК микобактерий в процессе ее репликации. По данным H. Kasai и соавт. (2000), ген gyrB является отличной мишенью для молекулярного определения вида микобактерий. В отличие от таких генов как 16S-23S rRNA, 16S-23S rRNA ITS, регион gyrB обладает большей вариабельностью нуклеотидной последовательности и вследствие этого более высокой видоспецифичностью. Анализ гена gyrB позволяет, помимо идентификации тех видов, которые можно определить и с помощью анализа нуклеотидных последовательностей вышеуказанных генов, дифференцировать M. kansasii от M. gastri, M. malmoense от M. szulgai, M. marinum от M. ulcerans, а также виды, входящие в M. tuberculosis complex, за исключением M. bovis BCG.
На сайте GenBank были взяты нуклеотидные последовательности гена gyrB из генома разных видов микобактерий. С помощью программы Vector NTI 10, AlignX (Invitrogen, США) проведено выравнивание нуклеотидных последовательностей ДНК разных видов микобактерий относительно друг друга.
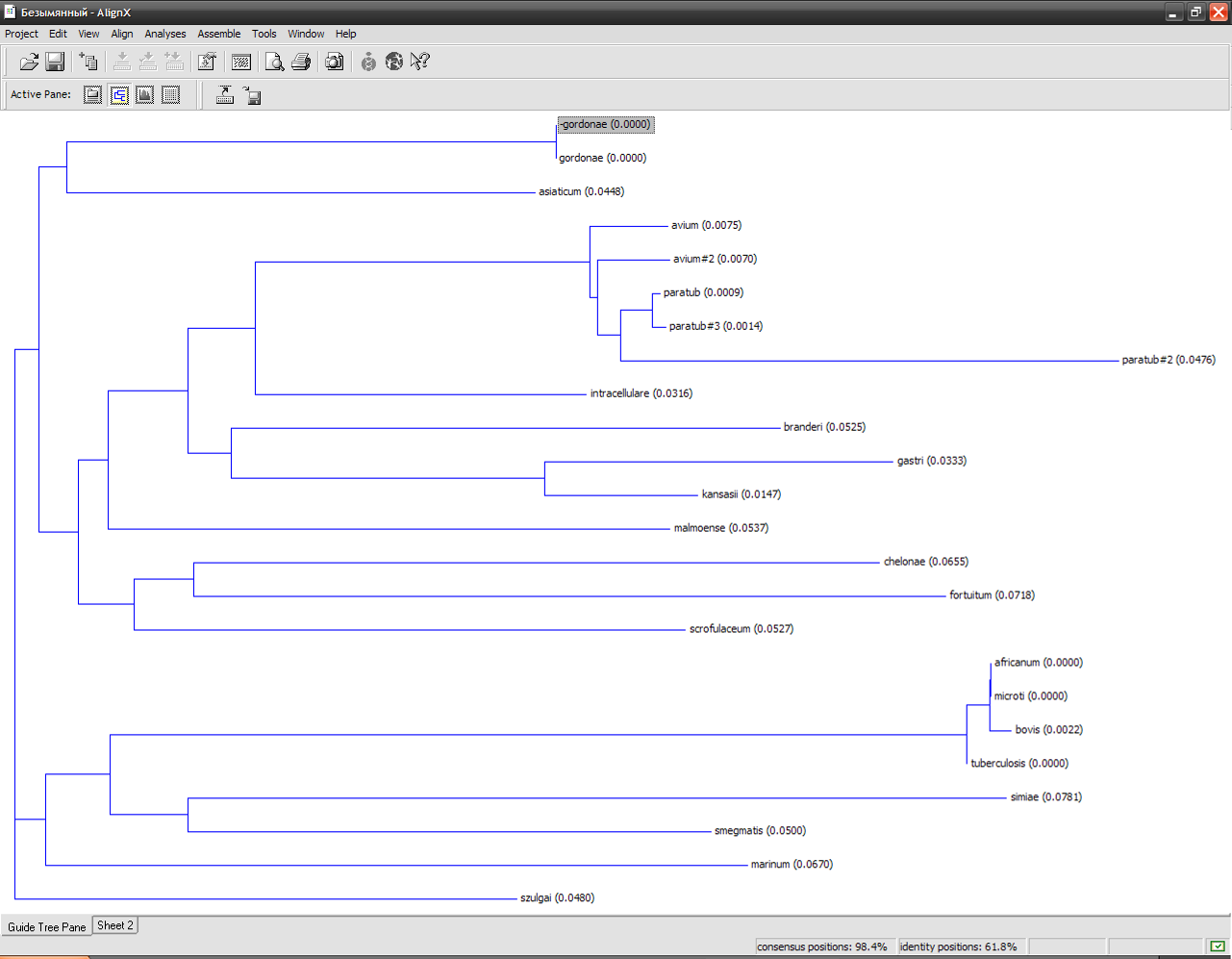
Выявлено, что ген gyrB представлен как консервативными областями, так и вариабельными видоспецифичными участками. Консервативные участки наиболее подходили для дизайна праймеров, а полиморфные вариабельные участки – это регион подбора видоспецифичных олигонуклеотидных зондов. В работе M. Fukushima и соавт. (2003) с целью видовой идентификации микобактерий предложено анализировать участок гена gyrB размером около 184 п.н. В свою очередь по результатам проведенного выравнивания нуклеотидных последовательностей этого гена стала очевидной целесообразность расширения данной области, которая в итоге составила около 400 п.н., тем самым, была повышена специфичность метода. Построение дендрограммы производили с помощью программы Vector NTI 10 (Invitrogen, США) и алгоритма Clustal W (рис.4).
При анализе дендрограммы, построенной для 20 видов микобактерий, установлено, что, выбранная область гена gyrB, подходит для создания тест-системы для видовой идентификации микобактерий, а именно «БИОЧИП IMS».
Микрочип «IMS» содержит 49 видоспецифичных дискриминирующих иммобилизованных олигонуклеотидов, 3 маркерные ячейки для позиционирования изображения и 20 резервных ячеек. Гелевые ячейки, содержащие видоспецифичные дискриминирующие олигонуклеотиды, объединены в 4 группы.