Семян и зародышей редких видов растений
| Вид материала | Автореферат диссертации |
- Красная книга, 61.32kb.
- Молекулярно-генетический анализ генофондов редких и исчезающих видов растений пермского, 701.05kb.
- Венции должны содействовать восстановлению исчезающих видов, в частности, путем разработки, 98.37kb.
- «Красной книге», 31.74kb.
- Школьные экскурсии, 230.88kb.
- Программа для поступающих в нввкус по специальности «Психология», 284.28kb.
- Международного Союза Охраны Природы Питеру Скотту. По его инициативе в мсоп в 1949, 1423.12kb.
- «Путешествие по регионам Большого Алтая, 177.44kb.
- Перечень видов продукции, на которые оформляются сертификаты качества муки, хлебобулочных, 13.78kb.
- Лекция Многообразие растение. Водоросли Систематика растений, 162.25kb.
4.Создание генетического банка in vitro.
В ходе проведенных исследований оптимизирована система размножения и сохранения in vitro редких и ценных представителей Iris, Belamcanda, Paeonia и Fritillaria с использованием культуры семян и зародышей (Рис.10).
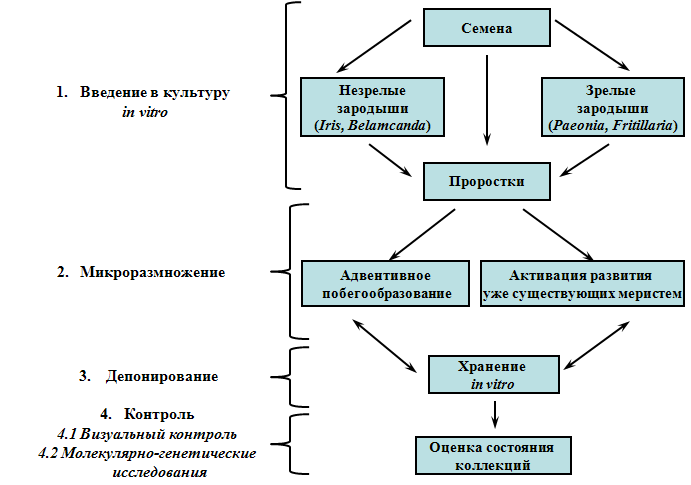
Рис.10. Cхема размножения и сохранения in vitro редких и ценных представителей Iris, Belamcanda, Paeonia и Fritillaria с использованием культуры семян и зародышей.


Рис.11. Стадия пролиферации I.ensata, P.anomala, F.ruthenica.
Подобраны и оптимизированы условия культивирования исследованных таксонов на всех основных этапах: введения в культуру, микроразмножения и длительного депонирования.
Создан генетический банк in vitro представителей родов Iris, Belamcanda, Paeonia и Fritillaria, в том числе включающий 37 образцов видов, занесенных в Красную книгу РФ.
5.Использование ISSR-анализа для идентификации и паспортизации растений в коллекциях генетических банков in vitro.
В последнее десятилетие наряду с традиционными приемами исследований все более широкое использование получают молекулярно-генетические методы, с применением которых, в настоящее время созданы ДНК-банки ценных, редких и исчезающих видов растений, проводятся исследования по изучению внутривидовой изменчивости сохраняемых объектов, уточнению спорных вопросов их систематики и классификации, разработке методики генетической паспортизации популяций и исследованию генетической стабильности хранящихся ex situ таксонов (Журавлев и др., 1999; Артюкова и др., 2001; Козыренко и др, 2004, 2009).
Семена некоторых представителей рода Iris, в частности редких видов подрода Iris (I.pumila, I.aphylla, I. scariosa, I. variegatа) и видов серии Spuriae подрода Xyridion (I.notha) (Родионенко, 1961) практически не отличимы по анатомо-морфологическим признакам, что затрудняет работу по созданию генетических банков in vitro, т.к. в спорных случаях на установление подлинности образцов может уйти (в зависимости от глубины покоя семян) до трех – пяти лет. Использование высокоэффективных молекулярных методов, таких как ISSR-анализ, дает возможность для быстрой и точной идентификации генетической подлинности образцов на родовом, видовом и популяционном уровне(Joshi et al, 2000; Боронникова, 2009; Wang et al, 2009), что может способствовать решению проблемы.
При проведении ISSR-анализа протестировали 14 ISSR-праймеров, из которых для дальнейшего анализа было отобрано 8, обеспечивающих получение достаточного количества полиморфных ампликонов.
Количество амплифицированных ISSR-фрагментов было различным: наименьшее их число – 13, было получено при использовании праймера М12, основанного на динуклеотидном повторе [CA], наибольшее – 27, для праймера 844В, в состав которого входит динуклеотидный повтор [CТ].
Размер амплифицированных ДНК-фрагментов варьировал в диапазоне от 125 п.н. (праймер М2) до 2290 п.н. (праймер 844В).
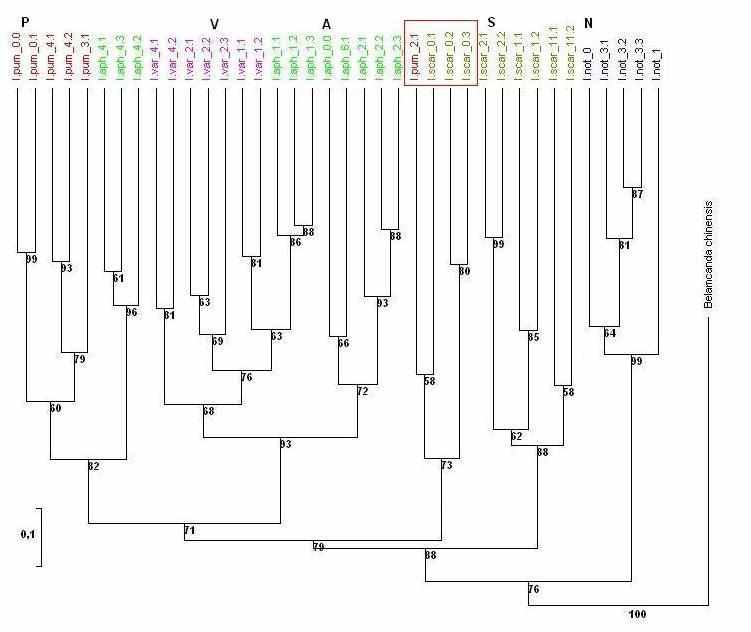
По полученным на основе ISSR-спектров данным были составлены бинарные матрицы и проведен кластерный анализ методом UPGMA (Рис.12).
□ – образцы неустановленной видовой принадлежности
Рис.12. Дендрограмма генетических взаимоотношений видов рода Iris, построенная методом NJ (Jaccard) по результатам ISSR-анализа. В узлах ветвей указан индекс бутстрэпа(%). Буквами обозначен контроль: P – I. pumila, N – I.notha, A – I. aphylla, S – I. scariosa, V – I. variegatа.
Установлено, что по степени генетической близости исследуемые образцы объединяются в 6 ( Belamcanda chinensis рассматривается как внешняя группа) основных кластеров, 5 из которых соответствуют отдельным рассматриваемым видам. На наибольшем генетическом расстоянии ото всех находится I. notha, что подчеркивает его обособленность, как вида, принадлежащего к другому, систематически удаленному, подроду Xyridion (серия Spuriae). Для остальных 4-х видов, показано, что результаты проведенного ISSR-анализа также, в целом, отражают представления современной систематики о генетическом родстве I. pumila, I. aphylla, I. scariosa и I. variegatа, входящих в состав подрода Iris (Родионенко, 1961, Mathew, 1981). Наиболее тесная связь внутри подрода наблюдается для I. aphylla и I. variegatа, несколько удален от них вид I. pumila и, наконец, самым большим генетическим расстоянием характеризуется I.scariosa.
Большинство из проанализированных образцов четко кластеризуются в соответствии с их видовой принадлежностью, однако некоторые образцы попадают в чужие кластеры, что указывает на их неверное определением. Так образцы I.aph_1 и I.aph_4, полученные из одного источника и включенные в работу как I. aphylla, по результатам кластерного анализа относятся к другим видам – I.variegatа и I.pumila соответственно. Также по данным кластерного анализа, проведенного на основе полученных ISSR-спектров и дополнительного анализа методом главных координат, для образцов I.scar_0 и I.pum_2, включенных в эксперимент как I.scariosa и I. pumilа, было показано, что данные образцы, образуют отдельный кластер и, очевидно, не принадлежат ни к одному из заявленных видов.
Однако по фенотипу исходные родительские растения являются заявленными I.scariosa и I.pumilа. В связи с чем, было выдвинуто предположение о возможном гибридном происхождения данных образцов. Для проверки этого предположения данные полученных ISSR-спектров всех образцов I. scariosa и I. pumilа по всем 8 праймерам были дополнительно проанализированы с помощью программы NewHybrids (Version 1.1 beta) (Anderson, 2003) (Табл.6).
Таблица 6
Анализ ISSR-профилей образцов I.pumila и I.scariosa в программе NewHybrids.
| Номер образца | Статистическая вероятность принадлежности образца к видовой/гибридной категории | |||||
| Pure_0 | Pure_1 | F1 | F2 | 0_Bx | 1_Bx | |
| 1* | 0.00000 | 0.95544 | 0.00237 | 0.00221 | 0.00002 | 0.03996 |
| 2 | 0.00000 | 0.84397 | 0.01990 | 0.00678 | 0.00002 | 0.12933 |
| 3 | 0.00000 | 0.99596 | 0.00001 | 0.00000 | 0.00000 | 0.00403 |
| 4 | 0.00000 | 0.98752 | 0.00006 | 0.00002 | 0.00000 | 0.01240 |
| 5 | 0.00000 | 0.99343 | 0.00010 | 0.00002 | 0.00000 | 0.00645 |
| 6 | 0.78334 | 0.00000 | 0.00000 | 0.00015 | 0.21651 | 0.00000 |
| 7 | 0.81214 | 0.00000 | 0.00000 | 0.00032 | 0.18754 | 0.00000 |
| 8 | 0.64074 | 0.00000 | 0.00000 | 0.00009 | 0.35917 | 0.00000 |
| 9 | 0.90982 | 0.00000 | 0.00000 | 0.00001 | 0.09017 | 0.00000 |
| 10** | 0.99988 | 0.00000 | 0.00000 | 0.00000 | 0.00012 | 0.00000 |
| 11 | 0.94268 | 0.00000 | 0.00000 | 0.00001 | 0.05731 | 0.00000 |
| 12 | 0.00031 | 0.00000 | 0.00024 | 0.02076 | 0.97869 | 0.00000 |
| 13 | 0.03063 | 0.00000 | 0.00000 | 0.00136 | 0.96801 | 0.00000 |
| 14 | 0.00183 | 0.00000 | 0.00017 | 0.01681 | 0.98119 | 0.00000 |
| 15 | 0.00083 | 0.00000 | 0.00009 | 0.01094 | 0.98814 | 0.00000 |
По полученным с помощью программы NewHybrids результатам, установлено, что все проанализированные генотипы данной выборки, кроме спорных, с высоким уровнем апостериорной вероятности – 84,4-99,5% и 64,1 – 99,9% для образцов I.pumilа и I.scariosa соответственно, отнесены программой к двум родительским видам. Что же касается спорных образцов I.scar_0 и I.pum_2, то с высокой долей апостериорной вероятности (96,8-98,8%) программа определяет их как бэккроссы к I.scariosa, что вполне объясняет наличие у исходных растений и полученных с них образцов семян фенотипических признаков двух видов одновременно. Для окончательного подтверждения или опровержения данной гипотезы и получения максимально достоверного результата необходимо проведение цитологических исследований и кариологического анализа с прямым подсчетом хромосом.
Проведенный ISSR-анализ позволил изучить генетическую изменчивость исследуемых образцов на межвидовом и популяционном уровне.
Всего в результате проведенного ISSR-анализа было получено и проанализировано 149 ДНК-фрагментов. Уровень полиморфизма варьировал внутри видов в широких пределах и в среднем составил: 69,60% для I. pumilа; 71,60% для I. notha; 77,36% для I. scariosa; 75,32% и 70,35% для I.variegata и I. aphylla соответственно и 53,2% для Belamcanda chinensis. (Табл. 7).
Таблица 7
Полиморфизм межмикросателлитных последовательностей исследуемых образцов.
| Вид / Праймер | Полиморфные ампликоны, % | |||||||
| 844B | UBC881 | M1 | M2 | M3 | М4 | M12 | M14 | |
| I.pumila | 80,25 | 71,43 | 81,25 | 30,00 | 91,66 | 78,57 | 77,78 | 46,15 |
| I.notha | 84,61 | 66,67 | 72,22 | 63,64 | 63,64 | 85,71 | 66,67 | 66,67 |
| I.scariosa | 80,00 | 84,61 | 88,24 | 57,14 | 92,31 | 77,78 | 83,33 | 57,14 |
| I.variegata | 85,71 | 94,12 | 73,33 | 50,00 | 66,67 | 78,57 | 87,50 | 66,67 |
| I.aphylla | 92,31 | 75,00 | 83,33 | 66,67 | 70,00 | 60,00 | 55,56 | 60,00 |
| Iris | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 94,74 |
| Belamcanda chinensis | 50,00 | 63,64 | 64,29 | 75,00 | 40,00 | 40,00 | 62,50 | 30,00 |
| Все образцы | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 |
По полученным экспериментальным данным у всех исследованных видов были выявлены общие и специфические мономорфные и полиморфные ДНК-фрагменты. Наличие мономорфных фрагментов у близких видов предполагает общность структурно-функциональной организации их геномов. Для 5 видов рода Iris с помощью праймера М14 показано наличие одного общего специфичного мономорфного фрагмента длинной 390 п.н. (Рис.13), а для Belamcanda – четырех: 510 п.н., 1075 и 310 п.н., 1325 п.н. – праймеры М3, М4 и М12 соответственно Мономорфные ДНК-фрагменты могут быть обозначены как «родовые» и считаться ISSR-маркерами для данных представителей родов Iris и Belamcanda.
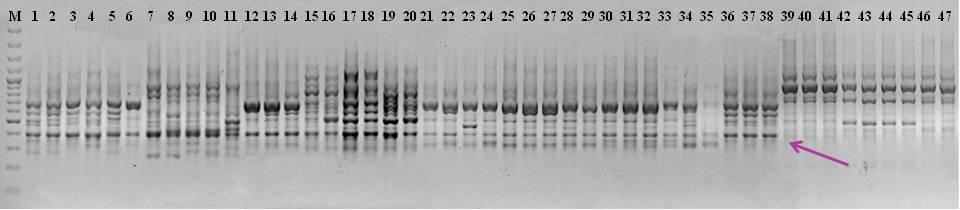
 мономорфный для видов рода Iris ДНК-фрагмент.
мономорфный для видов рода Iris ДНК-фрагмент.Рис.13. ISSR-профили образцов, полученные при использовании праймера М14. Условные обозначения: М – маркер молекулярных размеров; 1-6 – I. pumila; 7-11 – I. notha; 12-20 – I.scariosa; 21-27– I. variegatа; 28-38 – I. aphylla; 39-47 – Belamcanda chinensis.
В результате проведенного исследования было выявлено несколько специфичных «видовых» ISSR-маркера. Наименьшее их число – 1 – было получено в профилях образцов I. pumila (930 п.н. – праймер М4). Наибольшим числом видоспецифичных фрагментов характеризовались ISSR-спектры исследованных образцов Belamcanda chinensis и I. notha – 6 и 4 ДНК-фрагмента соответственно. На ISSR-спектрах I. scariosa было выявлено 3 видоспецифичных фрагмента. Для остальных исследованных видов подобных фрагментов детектировано не было. Подобные результаты можно объяснить систематической отдаленностью Belamcanda chinensis и I. notha, от видов подрода Iris и относительно небольшим количеством протестированных ISSR-праймеров.
Помимо видоспецифичных маркеров в ISSR-спектрах были обнаружены уникальные полиморфные ДНК-фрагменты, присущие только отдельным популяциям на уровне вида.
Выявленные в ходе данной работы специфичные «видовые» и «популяционные» полиморфные ISSR-маркеры позволяют использовать полученные данные в дальнейших исследованиях по разработке генетической паспортизации коллекций in vitro редких и исчезающих видов растений с использованием существующих современных методик (Соболев и др, 2006; Гончарова и др., 2009; Боронникова, 2009).
В данной работе изучалась возможность использования метода молекулярно-генетического маркирования трех популяций Belamcanda chinensis, основанного на ISSR-анализе (Боронникова, 2009), в котором учитываются как мономорфные, так и полиморфные ДНК-фрагменты, характеризующие геномы изучаемых объектов на надвидовом, видовом и популяционном уровнях.
Из всех полученных в ходе ISSR-анализа ампликонов для паспортизации объектов было отобрано 22 четко воспроизводимых мономорфных и полиморфных ДНК-маркера.
Род Belamcanda является монотипным в связи с чем, исходная методика паспортизации была несколько модифицирована и для используемых в маркировании ампликонов были приняты следующие обозначения: BG – мономорфные и BP – полиморфные ISSR-маркеры, специфичные для вида; BS – полиморфные ISSR-маркеры, специфичные для отдельных популяций. Для каждой из трех исследованных популяций было отобрано 9 фрагментов, сочетания которых уникальны и не совпадают ни у одной из них (Табл.8).
Таблица 8.
Молекулярно-генетические формулы популяций Belamcanda chinensis.
| Популяция | Тип фрагментов-ДНК | ||
| BG | BP | BS | |
| В.5 | BG310М4 BG595М3 BG1325М12 | BP635М4 ; BР720М1 ; BP805М2 | BS490М3 ; BS970М1 ; BS1080844В |
| В.6 | BP380М3 ; BP635М4 ; BP1185U881 | BS730М4 ; BS950М12 ; BS1030М2 | |
| В.8 | BP430М14 ; BP720М1 ; BP960М2 | BS365М4 ; BS1125U881 ; BS1660М12 | |
Помимо буквенно-цифровой записи паспорт популяции может быть представлен в виде штрих-кода (Рис.14).
| Маркер молекулярного веса | Штрих-код | Номер фрагмента | Обозначение ДНК-фрагмента |
| | | | |
| 1700 | | | |
| 1600 |  | 1 | BS1660М12 |
| 1500 | | | |
| 1400 | | | |
| 1300 |  | 2 | BG1325М12 |
| 1200 |  | 3 | BS1125U881 |
| 1100 | | | |
| 1000 | | | |
| 900 |  | 4 | BP960М2 |
| 800 | | | |
| 700 |  | 5 | BP720М1 |
| 600 |  | 6 | BG595М3 |
| 500 | | | |
| 400 |  | 7 | BP430М14 |
| 300 |   | 8 9 | BS365М4 BG310М4 |
Рис. 14. Молекулярно-генетический штрих-код популяции Belamcanda chinensis – В.8.
Предложенные генетические паспорта позволяют идентифицировать принадлежность объекта не только к роду (виду), но и к группе популяций, и к конкретной популяции, что дает возможность использовать их в качестве основы для дальнейших всесторонних исследований по молекулярному маркированию на популяционном уровне как Belamcanda chinensis, так и других редких таксонов, сохраняемых в коллекциях in vitro.
