Днк наномеханические роботы и вычислительные устройства
| Вид материала | Документы |
СодержаниеВзаимные преобразования между молекулами, осуществляющиеся при помощи Type I DNA topoisomerase [74]. |
- Программа-минимум кандидатского экзамена по специальности 05. 12. 13 «Системы, сети, 121.7kb.
- Программа-минимум кандидатского экзамена по специальности 05. 12. 13 «Системы, сети, 151.82kb.
- Домашние роботы, 181.38kb.
- Программа-минимум кандидатского экзамена по специальности 05. 12. 14 «Радиолокация, 134.92kb.
- Программа-минимум кандидатского экзамена по специальности 05. 12. 14 «Радиолокация, 236.33kb.
- Концепция: техники активации ДНК скрытые (виртуальные) структуры днк: множественные, 1618.54kb.
- В. А. Климёнов 2010 г. Рабочая программа, 267.99kb.
- Международная конференция «Microcad-2011», секция «Информатика и моделирование», 11.06kb.
- Учебно-методический комплекс дисциплины вычислительные системы, сети и телекоммуникации, 338.43kb.
- Определение: генетический код это система записи информации о последовательности расположения, 51.8kb.
Литература
- Adleman L.M. Molecular computation of solutions to combinatorial problems // Science. 1994. V.266, 1021-1024.
- Lipton R.J. Speeding up computations via molecular biology. Technical report, Princeton University, 1994.
- Lipton R.J. Using DNA to solve NPcomplete problems. Technical report, Princeton University, 1995.
- Pudlak P. Complexity theory and genetics. In: Proceedings of 9th Conference on Structure in Complexity Theory, 1994. P.183-195.
- Попов В.Ю. Полугрупповые модели процесса рестрикции. Международная алгебраическая конференция, посвященная 250-летию Московского университета и 75-летию кафедры высшей алгебры. Тезисы докладов. Москва, 2004. С.108.
- Schasfoort R., Schlautmann S., Hendrikse S., van den Berg A. Field-effect flow control for microfabricated fluidic networks // Science. 1999. V.286. P.942-945.
- Ni J., Zhong C., Coldiron S., Porter M. Electrochemically actuated mercury pump for fluid flow and delivery // Analytical Chemistry. 2001. V.73. P.103-110.
- Gallardo B., Gupta V., Eagerton F., Jong L., Craig V., Shah R., Abbott N. Electrochemical principles for active control of liquids on submillimeter scales. // Science. 1999. V.283. P.57-60.
- Ichimura K., Oh S., Nakagawa M. Light-driven motion of liquids on a photoresponsive surface. // Science. 2000. V.288. P.1624-1626.
- Weiler J., Hoheisel J. Combining the preparation of oligonucleotide arrays and synthesis of high-quality primers // Analytical Biochemistry. 1996. V.243. P.218-227.
- Bras M., Cloarec J., Bessueille F., Souteyrand E., Martin J., Chauvet J. Control of immobilization and hybridization on DNA chips by fluorescence spectroscopy // Journal of Fluorescence. 2000. V.10. N3. P.247-253.
- Pease A., Solas D., Sullivan E., Cronin M., Holmes C., Fodor S. Light-generated oligonucleotide arrays for rapid DNA sequence analysis // Proc. Natl. Acad. Sci. USA. 1994. V.91. P.5022-5026.
- McGall G., Barone A., Diggelmann M., Fodor S., Gentalen E., Ngo N. The efficiency of light-directed synthesis of DNA arrays on glass substrates // J. Am. Chem. Soc. 1997. V.119. P.5081-5090.
- Case-Green S., Mir K., Pritchard C., Southern E. Analysing genetic information with DNA arrays // Current Opinion in Chemical Biology. 1998. V.2. P.404-410.
- Lipshutz R., Fodor S., Gingeras T., Lockhart D. High density synthetic oligonucleotide arrays // Nature genetics supplement. 1999. V.21. P.20-24.
- Le Proust E., Pellois J., Yu P., Zhang H., Gao X. Digital light-directed synthesis. A microarray platform that permits rapid reaction optimization on a combinatorial basis // Journal of Combinatorial Chemistry. 2000. V.2. P.349-354.
- Kelley S., Boon E., Barton J., Jackson N., Hill M. Single-base mismatch detection based on charge transduction through DNA // Nucleic Acids Research. 1999. V.27. P.4830-4837.
- Roget A., Livache T. In situ synthesis and copolymerization of oligonucleotides on conducting polymers // Mikrochimica Acta. 1999. V.131. P.3-8.
- Singh-Gasson S., Green R., Yue Y., Nelson C., Blattner F., Sussman M., Cerrina F. Maskless fabrication of light-directed oligonucleotide microarrays using a digital micromirror array // Nature Biotechnology. 1999. V.17. N10. P.974-978.
- McGall G., Labadie J., Brock P., Wallraff G., Nguyen T., Hinsberg W. Light-directed synthesis of high-density oligonucleotide arrays using semiconductor photoresists // Proc. Natl. Acad. Sci. USA. 1996. V.93. P.13555-13560.
- Kwiatkowski M., Fredriksson S., Isaksson A., Nilsson M., Landegren U. Inversion of in situ synthesized oligonucleotides: improved reagents for hybridization and primer extension in DNA microarrays // Nucleic Acids Research. 1999. V.27. N24. P.4710-4714.
- Sosnowski R., Tu E., Butler W., O’Connell J., Heller M.Rapid determination of single base mismatch mutations in DNA hybrids by direct electric field control // Proc. Natl. Acad. Sci. USA. 1997. V.94. P.1119-1123.
- Westin L., Xu X., Miller C., Wang L., Edman C., Nerenberg M. Anchored multiplex amplification on a microelectronic chip array // Nature Biotechnology. 2000. V.18. N2. P.199-204.
- Zong Q., Schummer M., Hood L., Morris D. Messenger RNA translation state: the second dimension of high-throughput expression screening // Proc. Natl. Acad. Sci. USA. 1996. V.96. P.10632-10636.
- Schultz S., Smith D., Mock J., Schultz D. Single-target molecule detection with nonbleaching multicolor optical immunolabels // Proc. Natl. Acad. Sci. USA. 2000. V.97. N3. P.996-1001.
- Adessi C., Matton G., Ayala G., Turcatti G., Mermod J., Mayer P., Kawashima E. Solid phase DNA amplification: characterisation of primer attachment and amplification mechanisms // Nucleic Acids Research. 2000. V.28. N.20. P.87.
- Lucas S., Harding M. Detection of DNA via an ion channel switch biosensor // Analytical Biochemistry. 2000. V.282. P.70-79.
- Vo-Dinh T., Alarie J., Isola N., Landis D., Wintenberg A., Ericson M. DNA biochip using a phototransistor integrated circuit // Analytical Chemistry. 1999. V.71. P.358-363.
- van Gijlswijk R., Zijlmans H., Wiegant J., Bobrow M., Erickson T., Adler K., Tanke H., Raap A. Fluorochrome-labeled tyramides: use in immunocytochemistry and fluorescence in situ hybridization // The Journal of Histochemistry & Cytochemistry. 1997. V.45. N3. P.375-382.
- Ichimura K. Photoalignment of liquid-crystal systems // Chemical Reviews. 2000. V.100. P.1847-1873.
- Rodolfa K.T. et al. Nanopipette paints DNA picture // Angew. Chem. Int. Ed. 2005. V.44.
- Drmanac R., Crkvenjakov R. 1987. Method of sequencing of genomes by hybridization of oligonucleotide probes. Yugoslav patent application 570/80.
- Drmanac R., Labat I., Brukner L., Crkvenjakov R. Sequencing of megabase plus DNA by hybridization: Theory and method // Genomics. 1989. V.4. P.114-128.
- Лысов Ю.П., Флорентьев В.Л., Хорлин А.А., Храпко К.Р., Шик В.В., Мирзабеков А.Д. Определение нуклеотидной последовательности ДНК гибридизацией с олигонуклеотидами. Новый метод // Доклады Академии Наук СССР. 1988.
- Bains W., Smith G.C. A novel method for nucleic acid sequence determination // J. Theor. Biol. 1988. V.135. P.303-307.
- Pevzner P.A. Computational Molecular Biology: An Algorithmic Approach. MIT Press, 2000.
- Pevzner P.A., Lipshutz R.J. Towards DNA sequencing chips // Symposium on Mathematical Foundations of Computer Science. 1994. LNCS. V.841. P.143-158.
- Pevzner P.A., Lysov Yu.P., Khrapko K.R., Belyavsky A.V., Florentiev V.L., Mirzabekov A.D. Improved chips for sequencing by hybridization // J. Biomol. Struct. Dyn. 1991. V.9. P.399-410.
- Preparata F., Frieze A., Upfal E. Optimal reconstruction of a sequence from its probes // Journal of Computational Biology. 1999. V.6. N34. P.361-368.
- Preparata F., Upfal E. Sequencing by hybridization at the information theory bound: An optimal algorithm // Journal of Computational Biology. 2000. V.7. N34. P.621-630.
- Shamir R. Algorithms in molecular biology: Lecture notes, 2001. tau.ac.il/ rshamir/algmb/algmb00.phpl.
- Bodlaender H.L., Downey R.G., Fellows M.R., Hallett M.T., Wareham H.T. Parameterized complexity analysis in computational biology // Computer Applications in the Biosciences. 1995. V.11. P.49-57.
- Попов В.Ю. Вычислительная сложность проблем, связанных с расшифровкой ДНК гибридизацией // Доклады Академии Наук. 2005. Т.403. N3. C.1-3.
- Попов В.Ю. О проблеме расшифровки ДНК гибридизацией // IX международная конференция "Интеллектуальные системы и компьютерные науки", 23-27 октября 2006, Москва. Т.1. С.216-217.
3. Материалы для создания ДНК наномеханических устройств
На сегодняшний день существует довольно много различных моделей устройств на основе ДНК. Достаточно сказать, что только различных моделей вычислительных устройств существует несколько десятков (см., например, [1] – [52], см. также библиографию в [53], [54]). Значительная часть этих исследований основана на экспериментах, проводившихся с существенно различными устройствами из генетического материала (см. [1], [2], [5], [9], [10], [12], [24], [28] – [30], [32] – [34], [36], [49], [51]). При этом в большинстве случаев конструктивными элементами устройств являются либо молекулы, заимствованные из естественной среды, либо молекулы, специально синтезированные для этих устройств, либо некоторые комбинации того и другого. Однако, начиная с самой первой конструкции, предложенной в [1], отчетливо прослеживается, пусть и не всегда полностью осознанная, тенденция к созданию не монолитных устройств, а устройств, собранных из отдельных деталей, имеющих свою функциональную нагрузку. В частности, в основе устройства, предложенного в [1], лежали молекулы двух видов, кодирующие ребра и вершины графа, соответственно. Эти молекулы формировали множество правильных и неправильных решений, которое проходило три этапа фильтрации. Эта конструкция допускает широкие возможности модификации. При этом во многих случаях результат модификации будет легко предсказуем, что открывает путь к теоретическому анализу конструкции и компьютерному моделированию. В частности, по вполне прозрачному закону могут быть заменены типы молекул. Могут подвергнуться изменению и процедуры фильтрации. В результате чисто теоретического анализа конструкции [1] возникла, в частности, весьма интересная модель, предложенная в [8], получившая дальнейшее экспериментальное развитие в [28]. Хотя устройства, рассматриваемые в [1], [8] и [28], состоят из весьма простых молекул, а процедуры фильтрации в рамках экспериментов имели биохимическую природу, с модельной точки зрения об этих устройствах вполне можно говорить как о собранных из деталей. Соответственно, можно ставить вопрос о разработке деталей для подобных устройств.
Дальнейшим логическим развитием устройства из большого количества независимых элементов стала идея использования вместо отдельных молекул групп молекул, эмулирующих автомат. В рамках этой идеи была предложена модель универсального вычислителя (см. [2], см. также [4], [55], [56]), а также устройство, которое может быть использовано в медицинских целях для контроля синтеза белков [3]. Автомат состоит из большой группы отдельных молекул, каждая из которых выполняет свою специфическую функцию. Совокупность этих молекул реализует автомат с двумя состояниями, двоичным входом и 765 синтаксически различными программами. Часть молекул, входящих в состав автомата, являются ферментами, обеспечивающими автомат энергией для взаимодействия его частей. Универсальный вычислитель представляет собой генетическую жидкость, в которую помещено большое количество одинаковых автоматов. На 1 мл генетической жидкости приходится 3 × 1012 автоматов, которые производят 6.6 × 1010 операций в секунду с точностью 99.9%. Хотя 0.1% - это сравнительно небольшая погрешность, для рассматриваемого вычислителя эта погрешность дает 6.6 × 107 ошибок на 1 мл за секунду.
Одной из основных причин столь высокой погрешности при вычислениях является слишком большое количество различных конструктивных элементов в автомате, между которыми не установлены достаточно надежные связи. С этой точки зрения существенно более предпочтительно выглядит идея использования самоорганизующихся наноструктур, поскольку в рамках этой модели устройство конструируется не как набор независимых молекул, а как единый механизм, в котором молекулы объединены при помощи межнуклеотидных связей.
Идея использования самоорганизующихся структур для создания устройств была предложена в [20], а начало изучения таких структур было положено в работе [37]. А именно, в [37] была предложена идея создания искусственных молекул ДНК, которые являются стабильными (несклонными к разрушению или изменению формы) и могут быть объединены в стабильные молекулярные конструкции. Такие молекулы называют наноузлами, а получаемые из них конструкции – нанорешетками.
Стабильность наноузлов и их склонность к образованию стабильных молекулярных конструкций позволяют рассматривать наноузлы в качестве «фигурных кирпичей» и дает возможность выйти на принципиально новый уровень абстракции в исследованиях. В свое время обнаружение нуклеиновых кислот и аминокислот и понимание того, что все генетические соединения получены из них и свойства генетического материала зависят только от них, позволило рассуждать по модулю нуклеотидов и аминокислот, не вдаваясь в их химическое устройство. Соответственно появление наноузлов дает возможность вести рассуждения по модулю этих новых структур.
В работах [57] – [59] описаны исследования по наноузлам и нанорешеткам, не сохраняющим геометрическую форму. Благодаря исследованиям [60] – [70] удалось синтезировать нанорешетки, которые могут сохранять геометрическую структуру.
Исследования [71] – [78] направлены на построение нанорешеток с сложной топологической структурой и разработку технологий, позволяющих из одинаковых нуклеотидных последовательностей получать нанорешетки с различной топологией.
В частности, в рамках исследований, связанных с изменением топологической структуры последовательностей ДНК и РНК, обнаружены молекулы DNA topoisomerase, которые позволяют осуществлять такие изменения формы нанорешеток, которые невозможно осуществить без разрыва уже существующих связей (см. [74], [75]). К сожалению, на сегодняшний день нет какого-либо общего критерия, который позволял бы говорить о том, является ли та или иная последовательность ДНК топоизомеразой и если является, то какие топологические изменения она может стимулировать. Поэтому опираться в этом вопросе можно пока только на экспериментальные данные.
Известно, что одни и те же молекулы DNA topoisomerase могут осуществлять топологические преобразования в несколько различных топологических форм. В частности, все взаимные преобразования между молекулами, представленные на рисунке 1, осуществляются при помощи Type I DNA topoisomerase [74].
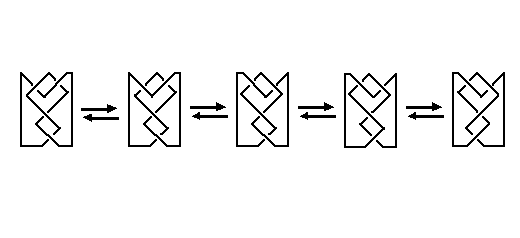
Рис.1. Взаимные преобразования между молекулами, осуществляющиеся при помощи Type I DNA topoisomerase [74].
В одной и той же молекуле может быть несколько различных видов DNA topoisomerase. При этом некоторые из топологических преобразований могут быть выполнены только молекулами фиксированного типа. Например, преобразование, представленное на рисунке 2, может быть осуществлено при помощи E. coli DNA Topoisomerase III, но его нельзя выполнить при помощи E. coli DNA Topoisomerase I (см. [75]).
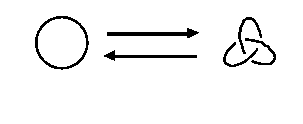
Рис.2. Топологическое преобразование, которое может быть осуществлено при помощи E. coli DNA Topoisomerase III, но нельзя выполнить при помощи E. coli DNA Topoisomerase I (см. [75]).
Следует отметить, что, хотя многие свойства биологических последовательностей можно получить из их линейной структуры посредством изучения отдельных участков и получения свойств отдельных генов, имеются примеры, указывающие на то, что это не всегда так. Весьма близкие последовательности могут выполнять совершенно разные функции (см., например, [79] – [83]). В то же время принципиально различные последовательности могут выполнять одинаковые функции (см., например, [84] – [88]). Одной из основных причин этого является то, что линейная структура биологических последовательностей не отражает их трехмерных свойств, которые несут достаточно важную смысловую нагрузку: на уровне трехмерных структур могут наблюдаться мутации (см., например, [89] – [91]); трехмерные структуры имеют непосредственное отношение к склонности и сопротивляемости болезням, в частности, это справедливо по отношению к болезни Альцгеймера [92]. Таким образом, исследования по использованию молекул DNA topoisomerase представляют интерес не только с точки зрения конструирования нанорешеток с заданными свойствами, но и для конструирования различных медицинских нанороботов. Кроме того, для нанотехнологий молекулы DNA topoisomerase представляют интерес как инструмент, позволяющий разрывать связи между нуклеотидами. В частности, это можно использовать при проектировании наноустройств на основе принципа свободной энергии.
Серия работ [93] – [104] направлена на создание хорошо масштабируемых нанорешеток с периодически повторяющимися свойствами. Для отдельных типов масштабируемых решеток с периодически повторяющимися свойствами [105] предложена технология, позволяющая произвольным образом модифицировать их базовую структуру [106]. В исследованиях [39], [107], [108] акцент сделан на создании нанорешеток со сложной нерегулярной структурой. В [109] проводились исследования по созданию нанорешеток, сохраняющих не только взаимное расположение нуклеотидов, но и заданную трехмерную форму, а также, по разработке технологий, позволяющих регистрировать трехмерную форму нанорешеток.
Другим важным строительным материалом для создания наномеханических устройств являются искусственно синтезированные молекулы, отдельные части которых обладают физическими свойствами различных молекул, что позволяет получить молекулу, способную проявлять свойства нескольких молекул одновременно (см., в частности, [110] – [116]). В частности, в [52] предложено устройство, конструктивными элементами которого являются и нанорешетки, и молекулы, проявляющие одновременно свойства ДНК и РНК.
Еще одно направление в области создания материалов для наномеханических устройств связано с использованием модифицированных нуклеотидов и аминокислот. У этого пути есть очевидное преимущество, заключающееся в расширении спектра доступных материалов и получении материалов с новыми свойствами, и не менее очевидный недостаток – мы не знаем новых свойств новых материалов. До некоторой степени с получением модифицированных или новых нуклеотидов и аминокислот помогает сама Природа. Хорошо устоявшуюся формулу, согласно которой в ДНК используются четыре вида нуклеотидов, в РНК используются четыре вида нуклеотидов, в белках используется 20 видов аминокислот, нельзя считать абсолютно верной, поскольку есть и другие виды нуклеотидов и аминокислот. Как и в большинстве «хороших» правил, в универсальном генетическом коде есть исключения. При этом под исключениями мы понимаем кодирование, являющееся стандартным для того или иного вида организмов, но отличающееся от универсального. Следует отметить, что кроме исключений могут наблюдаться также аномалии: изменения в коде, связанные с мутациями и механизмами их подавления. На сегодняшний день различных исключений в генетическом коде известно достаточно много. Наиболее типичные из них связаны с изменениями значений кодонов: кодоны могут кодировать другую аминокислоту; кодоны могут кодировать не только свою аминокислоту, но и начало считывания белка и т.д. Кроме исключений, связанных с изменением значений, есть и более серьезные. Например, кодон ACT при определенном контексте может быть не Stop-кодоном, а кодом аминокислоты селеноцистеин, которой в стандартном списке из двадцати аминокислот нет (см., например, [117], [118]). Большое количество исключений и мутаций наблюдается и для нуклеотидов. В частности, интересным объектом в этом отношении является транспортная РНК. Транспортная РНК состоят примерно из 70-100 нуклеотидов. Однако при определенных условиях транспортная РНК может выступать в качестве фрагмента в последовательности, состоящей из многих сотен нуклеотидов. Для считывания универсальной кодовой таблицы достаточно 31 разновидности транспортной РНК. Тем не менее даже у бактерий 45 различных транспортных РНК. Для их кодирования используется 78 генов. У дрожжей этих генов – около 400, у дрозофилы – около 750, у лягушки – около 8 000. Таким образом, Природа с большим запасом определяет не только количество различных видов транспортных РНК, но и количество способов их кодирования. Точное объяснение этой «щедрости» биологам еще предстоит найти. Однако это безусловно указывает на особую важность значения транспортной РНК для функционирования живых организмов, а также на высокую потребность в компенсации различных мутаций, наблюдающихся у транспортных РНК. Транспортная РНК считается наиболее изменчивой молекулой (см. [119], [120]). Причем изменениям подвержена не только последовательность нуклеотидов, но и сами нуклеотиды. Интересно отметить, что это свойство наблюдается практически для всех видов организмов вне зависимости от сложности их генома. Изменения в нуклеотидах для транспортной РНК настолько типичное явление, что для транспортной РНК имеет смысл рассматривать расширенный алфавит. Кроме четырех стандартных нуклеотидов A, U, C и G, в транспортной РНК используются еще четыре: dihydrouridine (D), pseudouridine (P), ribosylthymine (T), inosine (I). Также в транспортных РНК встречается группа нуклеотидов methylation с общим обозначением M. Встречаются и другие модифицированные нуклеотиды. Структура транспортной РНК достаточно хорошо изучена. В работе [121] построена модель транспортной РНК, считающаяся на сегодняшний день общепризнанной. Эта модель состоит из 76 нуклеотидов. Определенные позиции в транспортной РНК вне зависимости от ее разновидности всегда должны быть заняты нуклеотидами фиксированного вида (в противном случае молекула перестанет правильно функционировать). Имеется всего 27 таких позиций, некоторые из которых обязательно должны быть заняты измененными нуклеотидами. Таким образом, транспортная РНК не только существенно расширяет список доступных для использования нуклеотидов, но и указывает на то, что использование модифицированных нуклеотидов не обязательно препятствует использованию устройств в живых системах. Кроме того, в транспортной РНК можно наблюдать еще одно интересное свойство, связанное с принципом минимизации свободной энергии: некоторые измененные нуклеотиды обладают отрицательной свободной энергией, которая не только препятствует образованию слабой связи самим нуклеотидом, но и мешает соседям. Еще одним источником получения модифицированных нуклеотидов является использование стандартных нуклеотидов после обработки особыми ферментами. В частности, такие нуклеотиды могут потерять одну или несколько степеней свободы, что позволит им демонстрировать принципиально новые свойства (см., например, [54], пункт 1.2). Наконец, при необходимости можно использовать искусственно созданные новые типы нуклеотидов (см., в частности, [54], пункт 1.3). Отметим, что конструкция вычислительного устройства в работе [51] использует шесть типов нуклеотидов.
Хотя теоретические возможности нанобиологической робототехники говорят в пользу того, что в перспективе она способна заменить собой большинство из традиционных технологий, совершенно ясно, что переход на эту технологию может происходить только постепенно и вопрос об интеграции нанобиологических устройств с традиционными еще долго будет оставаться актуальным. Серьезным препятствием на пути построения интерфейсов между нанобиологическими устройствами и электронными является довольно низкая электропроводность молекул ДНК. Поэтому существенный интерес представляет разработка новых материалов, интегрирующих в себе генетический материал и проводящие материалы. Следует отметить, что исследования в этой области представляют интерес не только с точки зрения создания модифицированных молекул ДНК с высокими проводящими свойствами, но и для разработки новых технологий поатомной сборки металлических соединений и т.д. На сегодняшний день ведутся активные исследования в области взаимодействия генетических материалов с другими функциональными молекулами (см., например, [122] – [134]), в том числе исследуются вопросы взаимодействия нанорешеток с другими материалами (металлами, полупроводниками и др.) (см., например, [135] – [162]). Ведутся исследования в области создания интегрированных конструкций из молекул ДНК и проводящих материалов, например, карбоновых нанотуб (см. [163]). Наиболее интересной представляется технология металлизации молекул ДНК, поскольку она позволяет повысить проводимость самой молекулы ДНК, а не использует молекулы ДНК как связующий материал для проводников. Существует несколько подходов к металлизации ДНК. В частности, нанесение металла на поверхность [164], создание искусственных нуклеотидов, содержащих атомы металла (например, палладия или меди [165], [166]), размещение ионов цинка между нуклеотидами [167]. В первом эксперименте по металлизации [168] была получена функциональная связь ДНК с серебром для установления проводимости между золотыми электродами. Позднее эта технология была существенно улучшена и распространена на ряд других металлов и полупроводников (см., например, [169] – [174]).
