Курсовая работа студентки 3 курса Бабской Евгении Михайловны " Однонуклеотидные полиморфизмы ppar-зависимых генов. Зависимость количества полиморфизмов от древности гена"
| Вид материала | Курсовая |
СодержаниеТаксономические группы генов выборки. |
- Курсовая работа Студентки 2 курса до гр. 2 «Г» Шиляевой Татьяны Владиславовны Ассистент:, 535.22kb.
- Вопросы к экзамену по курсу «Молекулярная биология» (вечернее отделение), 36.64kb.
- Курсовая работа студентки 1 курса, вечернего отделения, 38.18kb.
- 10 ч 00 мин Открытие сессии стендовых докладов Центральный холл нии онкологии тнц, 89.99kb.
- Московский гуманитарно-экономический институт теория государства и права курсовая работа, 352.27kb.
- Курсовая работа студентки Iкурса вечернего отделения, 574.72kb.
- Курсовая работа студентки Iкурса вечернего отделения, 574.72kb.
- Курс 3 Группа 306 Семестр 6 задание на курсовую роботу студентки кравцовой Виктории, 195.83kb.
- Генная инженерия, 313.05kb.
- Контрольная работа для 10 класса по теме «Агрегатные состояния вещества», 10.46kb.
1 2
Таблица 4. Таксономические группы генов выборки. Гены были разбиты на группы в соответствии с возникновением организмов, в которых были найдены их ортологи. Наиболее древними гены считаются те, ортологи которых были найдены в амебах, наиболее новыми считаются те, ортологи которых ищутся только среди млекопитающих. Можно заметить, что в самых новых генах содержится максимальное число полиморфизмов (около 4,6%), а в самых древних и основное части оно колеблется около 0,5%. Это можно объяснить, с одной стороны тем, что древность гена подразумевает устоявшуюся функцию, для поддержания которой необходима стабильность и неподверженность спонтанным мутациям, приобретенные в ходе эволюции. Но с другой стороны, большая вариабельность последовательностей новых генов является платформой для возникновения новых модификаций белков, лиганд-связывающих сайтов, комплексных функций, выполняемых экспрессирующимися белками, – своеобразным локальным двигателем естественного отбора. 5. Выводы.
6. Заключение. Однонуклеотидные полиморфизмы исследованной выборки PPAR-зависимых генов подчиняются общим законам появления и распределения данных замен. Характерные нарушения данных генов, в связи с появлением полиморфизмов, зачастую приводят к развитию тяжелых заболеваний или другим отклонениям в работе организма. Важность изучения однонуклеотидных полиморфизмов неоспорима: новые методы детекции замен, исследование эволюции и фенотипических проявлений полиморфизмов могут стать основой для фармацевтических разработок и важных открытий в области исследования таких заболеваний, как диабет, болезнь Альцгеймера, рак и ожирение. Появление в настоящее время большого числа проектов, посвященных изучение генома человека, таких как HAPMAP (ссылка скрыта), а также баз данных полиморфизмов и многих других вспомогательных ресурсов, содержащих информацию о генетическом материале, значительно ускоряет процесс появления новых технологий в медицине и позволяет исследованиям подниматься на все более новые уровни. Список литературы. 1. Anthony J. Brookes. The essence of SNPs. Gene 234; 1999; 177–186 2. Joke Reumers, Lucia Conde, Ignacio Medina, Sebastian Maurer-Stroh, Joost Van Durme, Joaquin Dopazo, Frederic Rousseau and Joost Schymkowitz. Joint annotation of coding and non-coding single nucleotide polymorphisms and mutations in the SNPeffect and PupaSuite databases. Nucleic Acids Research, 2008, Vol. 36, Database issue 3. Michael A. Eberle , Mark J. Rieder, Leonid Kruglyak, Deborah A. Nickerson. Allele Frequency Matching Between SNPs Reveals an Excess of Linkage Disequilibrium in Genic Regions of the Human Genome. PLoS Genet 2(9): e142. DOI: 10.1371/journal.pgen.0020142 4. Stephen T. Sherry, Minghong Ward, and Karl Sirotkin. Use of Molecular Variation in the NCBI dbSNP Database. Hum Mutat 15:68–75, 2000. 5. 2001 Nature Publishing Group ссылка скрыта 6. Danielle G. Lemay and Daniel H. Hwang. Genome-wide identification of peroxisome proliferator response elements using integrated computational genomics. J. Lipid Res. 2006. 47: 1583–1587. 7. Hyoung Doo Shin, Byung Lae Park, Lyoung Hyo Kim, Hye Seung Jung, Young Min Cho, Min Kyong Moon, Young Joo Park, Hong Kyu Lee, and Kyong Soo Park. Genetic Polymorphisms in Peroxisome Proliferator–Activated Receptor – δ Associated With Obesity. Diabetes 53: 847–851, 2004 8. J. R. Kidd, Y. Matsubara, C. M. Castiglione, K. Tanaka, K. K. Kidd. The Locus for the Medium-Chain Acyl-CoA Dehydrogenase Gene on Chromosome 1 Is Highly Polymorphic. GENOMICS 6.89-93 (1990) 9. Usha Varanasi, Ruiyin Chu, Qin Huang, Raquel Castellon, Anjana V. Yeldandi, and Janardan K. Reddy. Identification of a Peroxisome Proliferator-responsive Element Upstream of the Human Peroxisomal Fatty Acyl Coenzyme A Oxidase Gene. THE JOURNAL OF BIOLOGICAL CHEMISTRY Vol. 271, No. 4, Issue of January 26, pp. 2147–2155, 1996 10. Usha Varanasi, Ruiyin Chu, Su Chu, Rafael Espinosa, M. M. Lebeau, J. K. Reddy. Isolation of the human peroxisomal acyl-CoA oxidase gene: Organization, promoter analysis, and chromosomal localization . Biochemistry, April 1994. 11. Wei-Shiung Yang, Lee-Ming Chuang. Human genetics of adiponectin in the metabolic syndrome. J Mol Med (2006) 84: 112–121. 12. Kevin P. O'Brien, Maido Remm1 and Erik L. L. Sonnhammer. Inparanoid: a comprehensive database of eukaryotic orthologs. Nucleic Acids Research, 2005, Vol. 33. 7. Приложение. 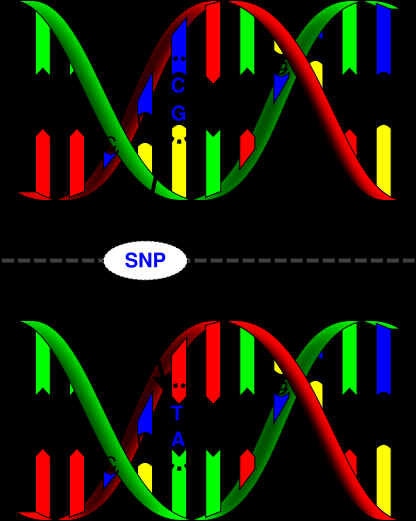 Рис.1 Однонуклеотидный полиморфизм. На рисунке приведен пример замены основания тимина на цитозин, приводящей к замене комплементарного основания. Выделено однонуклеотидное различие в последовательностях ДНК. (David Hall, 2007-07-06). . 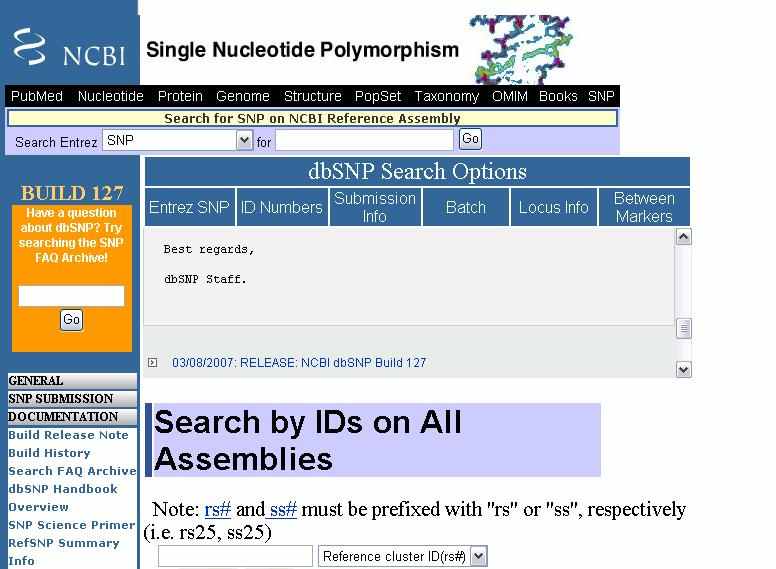 Рис 2. Главная страница базы данных dbSNP (ссылка скрыта). Записи базы содержат информацию о последовательности вокруг полиморфизма, описание популяции, содержащей вариацию и часто информацию о генотипе популяции или индивида. Поиск проводится по идентификатору гена для выдачи всех содержащихся в нем полиморфизмов, либо по id отдельного интересующего полиморфизма. 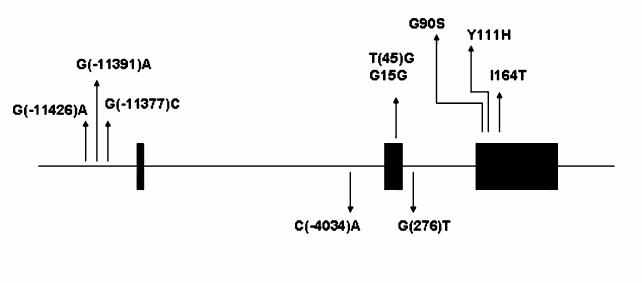 Рис 3. Структура гена адипонектина человека и распределение обычных полиморфизмов и редких мутаций, описанных в исследованиях генетических связей данного гена [11]. 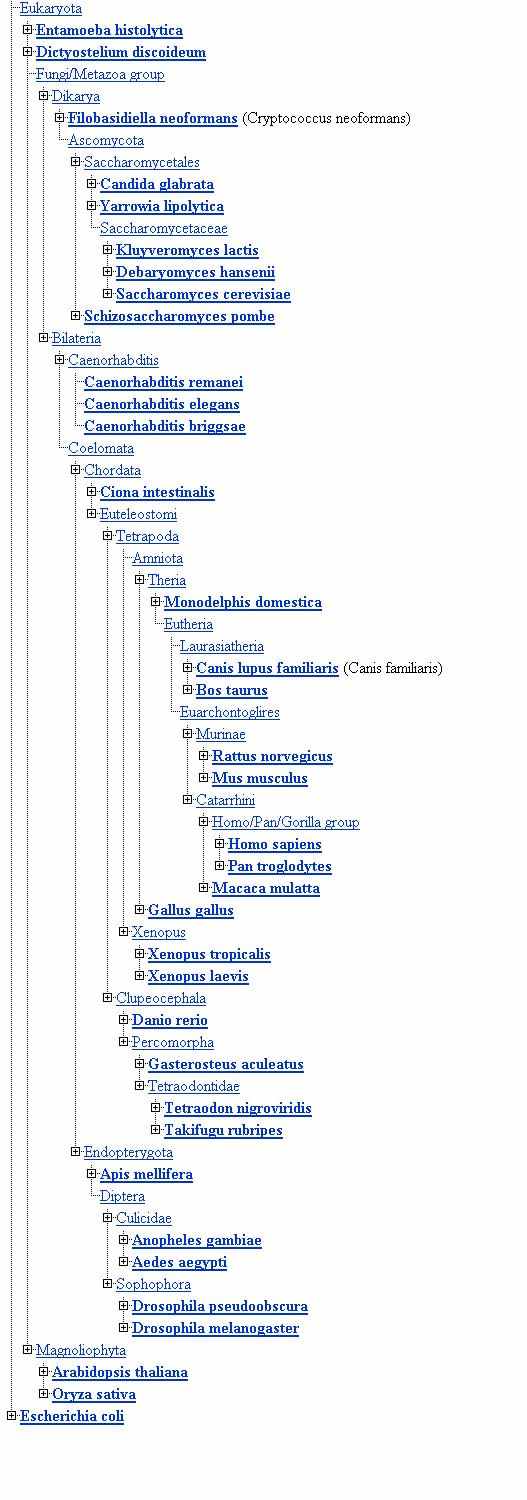 Рис.4. Таксономическое дерево видов, в которых были обнаружены ортологи исследуемых генов, полученное при помощи сервера NCBI, Taxonomy Entrez (ссылка скрыта).
Таблица 2. Количественные характеристики однонуклеотидных полиморфизмов выборки PPAR-зависимых генов.
Таблица 3. Характеристика полиморфизмов в выборке PPAR-зависимых генов. Данные для таблицы получены в базе данных dbSNP и разбиты на категории в связи с их особенностью: полиморфизмы, приводящие к аминокислотной замене, к замене аминокислоты на стоп-кодон, синонимичной замене, полиморфизмы 5’-концевой и 3’-концевой нетранслируемых областей мРНК, расположенные около 5’ и 3’-концов, приводящие к сдвигу рамки и полиморфизмы в интронах генов. |
