М. В. Ломоносова Факультет Биоинженерии и Биоинформатики Изучение регуляции азотофиксации в различных видах архебактерий Курсовая
| Вид материала | Курсовая |
- Курсовая работа студентки 3 курса Бабской Евгении Михайловны " Однонуклеотидные полиморфизмы, 334.26kb.
- М. В. Ломоносова Суббота, 9 октября Лекции, 198.89kb.
- «Эволюция числа ядрышкообразующих районов хромосом у животных», 467.26kb.
- Факультет биоинженерии и биоинформатики расписание осеннего семестра 5 курса 2009/2010, 58.13kb.
- Xi международная научная конференция студентов, аспирантов и молодых учёных, 268.04kb.
- Темы курсовых работ для студентов 1 2-го курсов факультета биоинженерии и биоинформатики, 17.82kb.
- Факультет Биоинженерии и Биоинформатики Построение трёхмерных структур апоформ флуоресцентных, 430.97kb.
- М. В. Ломоносова Факультет политологии Кафедра государственной политики Курсовая, 6kb.
- М. В. Ломоносова географический факультет кафедра мирового хозяйства Курсовая, 476.29kb.
- М. В. Ломоносова факультет психологии кафедра общей психологии программа, 128.66kb.
e) Methanococcoides burtonii
glnA

glnB

amt

Рис.6. Продолжение
f) Methanosarcina acetivorans
nifI2 vnfH nifI1 nifI2 vnfG vnfK vnfK vnfE vnfN















 nifH nifI1 nifI2 nifD nifK nifE nifN
nifH nifI1 nifI2 nifD nifK nifE nifN glnK amtB amtB



amt glnB


antK anfG anfD nifI1 nifI2





glnA

vnfE vnfN nifH



g) Methanopyrus kandleri

 MK0337 glnA
MK0337 glnA nifH

nifD

AmtB MK0054


 glnB
glnB h) Archaeoglobus fulgidus
amt2 glnB3 amt3 glnB2




amt1 glnB1


glnA

В процессе изучения оперонной структуры генов азотофиксации было обнаружено большое число дупликаций. Эволюцию дуплицирующихся генов в пределах исследуемых организмов можно последить по филогенетическим деревьям, построенным для каждой группы дуплицировавшихся генов в отдельности.
Многочисленные дупликации наблюдаются для генов, кодирующих субъединицы транспортера аммония (amtB) и регуляторные белки семейства РII: glnB, glnK, nifI1, nifI2 (Рис.7-8).
Рис. 7. Филогенетическое дерево AmtB белков-транспортеров аммония.
Условные обозначения: MTH-Methanobacterium thermoautotrophicum, MMZ-Methanosarcina mazei, MJ-Methanocaldococcus jannaschii, MA-Methanosarcina acetivorans, MK-Methanopyrus kandleri, MM-Methanococcus maripaludis, MCB-Methanococcoides burtonii, AF-Archaeoglobus fulgidus, MB-Methanosarcina barkeri. Зеленым отмечены гены, в промоторных областях которых обнаружены регуляторные сигналы.

Рис.8. Филогенетическое дерево белков семейства PII. Условные обозначения: MTH-Methanobacterium thermoautotrophicum, MMZ-Methanosarcina mazei, MJ-Methanocaldococcus jannaschii, MA-Methanosarcina acetivorans, MK-Methanopyrus kandleri, MM-Methanococcus maripaludis,MCB-Methanococcoides burtonii, AF-Archaeoglobus fulgidus, MB-Methanosarcina barkeri. Зеленым отмечены гены, в промоторных областях которых обнаружены регуляторные сигналы.
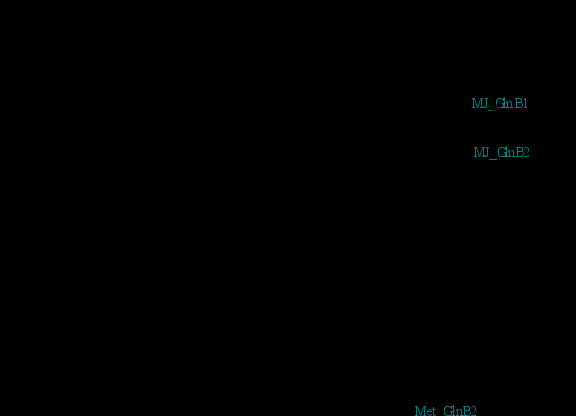
В геноме M.acetivorans дупликации на раннем этапе эволюции и последующей модификации подверглись гены, кодирующие субъединицы нитрогеназы, в результате чего в организме присутcтвуют три типа нитрогеназ:
- молибден-зависимая нитрогеназа NifHDKEN
- ванадий-зависимая нитрогеназа VnfHDKEN
- нитрогеназа AnfDK, не содержащая ни молибденового , ни ванадиевого кофактора Аннотация генов vnf и anf была проведена ранее биоинформатическими методами. (Galagan et al, 2002).
Филогенетическое дерево для белков, являющихся составными частями нитрогеназы, представлено на Рис.9.
Рис. 9. Филогенетическое дерево nif генов, кодирующих субъединицы нитрогеназы.
Условные обозначения: MTH-Methanobacterium thermoautotrophicum, MMZ-Methanosarcina mazei, MJ-Methanocaldococcus jannaschii, MA-Methanosarcina acetivorans, MK-Methanopyrus kandleri, MM-Methanococcus maripaludis, MCB-Methanococcoides burtonii, AF-Archaeoglobus fulgidus, MB-Methanosarcina barkery. Зеленым отмечены гены, в промоторных областях которых обнаружены регуляторные сигналы.
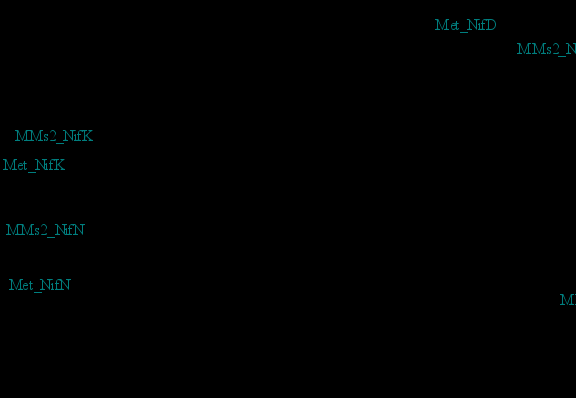
Интересно, что в первых двух случаях полностью сохраняется структура оперонов, кодирующих субъединицы денитрогеназ, что наводит на мысль о происхождении их путем дупликации первоначального оперона (Рис.6f).
Белки регуляторного каскада.
В археях отсутствуют все гены, кодирующие белки регуляторного каскада, характерного для бактерий, кроме генов, кодирующих белки семейства РII (Dixon and Kahn, 2004). Это объясняется принципиально различным механизмом регуляции у архей и эубактерий. В то время, как в бактериях экспрессия генов азотофиксации регулируется по принципу активации энхансер-связывающими белками, в археях эта регуляция осуществляется по принципу репрессии при помощи NrpR белка.
Известно, что белки РII семейства необходимы для быстрой посттранскрипционной регуляции азотофиксации, которая защищает клетку от затрат энергии, когда концентрация внутриклеточного аммония слишком высока (Kessler et al, 2002). В бактериях посттранскрипционная регуляция азотофиксации осуществляется за счет ковалентного ADP-рибозилирования железосодержащего белка, входящего в состав нитрогеназы. К сожалению, неизвестно, существует ли подобный механизм регуляции в археях, однако для M.maripaludis было показано, что для подобной регуляции необходимо присутствие не только GlnB и GlnK, как в бактериях, но и двух других членах этого семейства NifI1 и NifI2. Наличие этих двух белков в геноме характерно только для архей. ADP-рибозилирование железосодержащего белка нитрогеназы для архей не было установлено (Dixon and Kahn,2004).
В то время как GlnB и GlnK близки по структуре к бактериальным белкам семейства PII, что выражается в наличие сайта уридинилирования и строении Т-петли - специфической последовательности, отвечающей за связывание с эффектором (Yiang et al, 1997). NifI1 и NifI2 не имеют сайта уридинилирования, а по строению Т-петли отличаются от других гомологов и между собой (Kessler et al, 2002). Все это указывает на то, что механизм посттранскрипционной регуляции в археях происходит иначе, чем в организме бактерий.
Нами было показано,что гены nifI1 и nifI2 располагаются в одном опероне с генами субъединиц нитрогеназ, nif опероне. Исходя из этого, можно предположить, что они необходимы для посттранскрипционной регуляции фиксации молекулярного азота. Гены amtB, glnB и glnK также образуют один оперон, из чего можно сделать предположение, что GlnB и GlnK участвуют в регуляции транспорта аммония через плазматическую мембрану. Мы предполагаем, что при увеличении внутриклеточной концентрации глутамина, а значит, связанного азота, GlnB и GlnK находятся в активном состоянии и блокируют каналы-транспортеры аммония, продукты генов amt. При уменьшении же концентрации аммония уридилтрансфераза уридинилирует GlnB и GlnK, в результате чего они теряют способность к связыванию с каналом (Рис.10).











Аммоний
Р
 ис.10. Регуляция транспорта
ис.10. Регуляция транспорта аммония через плазмати-
ч
 ескую мембрану клетки. AmtB
ескую мембрану клетки. AmtBП
 ри увеличении концентра-
ри увеличении концентра-ции внутриклеточного
а
 зота “свободный” регу-
зота “свободный” регу- лятор закрывает канал-транс-
п
 ортер. При низкой - уридил- Концентрация глутамина
ортер. При низкой - уридил- Концентрация глутамина т
 рансфераза инактивирует
рансфераза инактивирует регулятор, который теперь
не может связываться с каналом.



 Уридилтрансфераза UMP
Уридилтрансфераза UMPGlnB или
GlnK
Поиск потенциальных регуляторных сайтов
Ранее биоинформатическими методами был определен регуляторный сигнал для NrpR, представляющий собой палиндром длиной18 нуклеотидов. Сайты такого вида были обнаружены пред генами, участвующими в азотофиксации, в геномах M.thermoautotrophicum, M.jannaschii и M.maripaludis. (Gelfand et al, 2000). Для того, чтобы более полно охарактеризовать структуру данного регуляторного сигнала, нами была построена диаграмма логo на основе всех сайтов, найденных перед генами азотофиксации в этих трех геномах. Как видно из Рис.11, данный сайт представляет собой инвертированный повтор длиной 16 нуклеотидов и имеет спейсер из 6 нуклеотидов.(Рис.11) Этот результат отличается от полученного ранее экспериментальными методами, согласно которому регуляторный сигнал имеет строение GGTT-(6)-AACC (Kessler and Leigh, 1999), а так же не согласуется и с ранее полученными биоиформатическими данными, согласно которым этот сайт представляет собой палиндром из 18 нуклеотидов (Gelfand et al, 2000). Из составленной нами диаграммы лого видно, что крайние позиции не являются информационно значимыми. Поэтому есть основание предполагать, что регуляторный сигнал азотофиксации в геномах M.thermoautotrophicum, M.jannaschii, M.maripaludis представляет собой инвертированный повтор длиной 16 нуклеотидов с консенсусом mGGAAr-6-yTTTCCk.
Рис. 11. Лого для сайта связывания NrpR. По горизонтальной оси указан номер позиции сайта связывания, по вертикальной – информационное содержание позиции. Высота столбца пропорциональна информационному содержанию данной позиции, относительная высота каждой буквы соответствует частоте нуклеотида в данной позиции.

Поиск регуляторного сигнала в других геномах
На основании филогенетического дерева ортологов NrpR была выделена группа организмов, обладающих высокой степенью сходства ортологов, а, следовательно, и регуляторных сигналов. В эту группу вошли геномы M.mazei, M.acetivorans, M.burtonii, M.barkeri. Поскольку ортологи NrpR из этих организмов содержат ДНК-связывающий и предполагаемый димеризующий домены, то можно предположит, что общий механизм взаимодействия регулятора с ДНК не изменился. Из чего можно сделать вывод, что следует искать регуляторный сигнал палиндромного строения.
Первоначально нами была осуществлена попытка поиска предполагаемых сайтов с использованием метода генетического футпринтинга. На основании промоторных областей всех генов, предположительно относящихся к регулону азотофиксации, Было построено множественное выравнивание 5-областей всех генов, предположительно относящихся к регулону азотофиксации. Однако на нем не было обнаружено продолжительных консервативных последовательностей.
Исходя из предположения, что во всех исследуемых организмах сайт связывания NrpR имеет палиндромное строение, с помощью программы SignalX был произведен поиск палиндромов различной длины (от 12 до 18) в регуляторных областях генов, участвующих в фиксации азота. Однако нам не удалось найти потенциальных сигналов, которые бы сохранялись перед ортологичными генами в разных геномах.
Взаимодействие NrpR-ДНК происходит путем связывания с плечами палиндрома НТН-доменов димера. Другие два домена, связываясь с соответствующими доменами второй субъединицы, образуют “мостик” между НТН-доменами над спейсерным участком регуляторного сигнала. В регуляторах исследуемой нами группы сохраняется только один ДНК-несвязывающий домен, что могло привести к укорочению длины “мостика”, а соответственно и спейсера. Таким образом, предполагая, что консенсус плечей палиндрома являетсясходным в исследуемых геномах, а длина спейсера изменяется, мы составили консенсус на основании уже известного регуляторного сигнала. Изменяя длину спейсера от 6 до 0 мы проводили поиск в исследуемых геномах, однако не удалось обнаружить потенциальных сигналов, сохраняющихся в разных геномах перед ортологичными генами.
Выводы:
- Проведено исследование регуляции азотофиксации в различных группах архей с помощью методов сравнительной геномики.
- Обнаружен ряд гомологов регуляторного белка NrpR. Показаны отличия в строении гомологов, косвенно указывающие на возможные различия в механизме регуляции азотофиксации.
- Обнаружены множественные дупликации генов, кодирующих субъединицы транспортера аммония (amt) и белки, регулирующие транспорт аммония (glnB, glnK).
- В геноме M.acetivorans обнаружены дупликации генов, кодирующих субъединицы нитрогеназы.
- На основании анализа оперонной структуры в различных геномах выдвинута гипотиза о роли различных белков PII семейства в регуляции азотофиксации.
- Подтверждена структура регуляторного сайта в геномах M.thermoautotrophicum, M.jannaschii и M.maripaludis.
- В других геномах обнаружить потенциального регуляторного сайта не удалось.
Список литературы.
Altschul S.F, Madden T.L., Schaffer A.A., Zhang J., Zhang Z., Miller W. and Lipman D.L.. (1997) Gappend BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Research, 1997, Vol. 25, No. 17: 3389-3402
Aravind L. and Koonin E.V. (1999) DNA-binding proteins and evolution of transcription regulation in the achaea. Nucleic Acids Research, Vol. 27, No. 23: 4658-4670
Atchley W.R., Rosinski J.A. (1999) Molecular evolution of helix-tern-helix proteins. J Mol Evol 49: 301-309
Bell S.D. and Jackson S.P. (1998) Transcription in Archaea. In “Mechanisms of transcription”, Cold Spring Harbor Laboratory Press.
Bult C.J., White O., Olsen G.J., Zhou L., Fleischmann R.D., Sutton G.G., Blake J.A., FitzGerald L.M., Clayton R.A., Gocayne J.D., Kerlavage A.R., Dougherty B.A., Tomb J.F., Adams M.D., Reich C.I., Overbeek R., Kirkness E.F., Weinstock K.G., Merrick J.M., Glodek A., Scott J.L., Geoghagen N.S., and Venter J.C. (1996) Complete genome sequence of the methanogenic archaeon, Methanococcus jannaschii. Science. 273 (5278): 1058-1073.
Cohen-Kupiec R., Blank C., and Leigh J.A. (1997) Transcriprional regulation in Archaea: In vivo demonstration of a repressor binding site in a methanogen. Proc. Nacl. Acad. Sci. 94: 13475
Crooks G.E., Hon G., Chandonia J.-M., and brenner S.E.. (2004) WebLogo: a sequence logo generator. Cold Spring Harbor Laboratory Press ISSN 1088-9051/04 14:1188-1190
Deppenmeier U., Johann A., Hartsch T., Merkl R., Schmitz R.A., Martinez-Arias R., Henne A., Wiezer A., Baumer S., Jacobi C., Bruggemann H., Lienard T., Christmann A., Bomeke M., Steckel S., Bhattacharyya A., Lykidi A., Overbeek R., Klenk H.P., Gunsalus R.P., Fritz H.J., and Gottschalk G. (2002) The genome of Methanosarcina mazei: evidence for lateral gene transfer between bacteria and archaea. J Mol Microbiol Biotechnol. 4 (4): 453-461.
Dixon R. and Kahn D.. (2004) Genetic regulation of biological nitrogen fixation. Nature Reviews, Microbiology, Vol. 2: 621
Doolittle W.F. and Brown J.R. (1994) Tempo, mode the progenote, and the universal root. Proc. Natl. Acad. Sci 91: 6721
Galagan J.E., Nusbaum C., Roy A., Endrizzi M.G., Macdonald P., FitzHugh W., Calvo S., Engels R., Smirnov S., Atnoor D., Brown A., Allen N., Naylor J., Stange-Thomann N., DeArellano K., Johnson R., Linton L., McEwan P., McKernan K., Talamas J., Tirrell A., Ye W., Zimmer A., Barber R.D., Cann I., Graham D.E., Grahame D.A., Guss A.M., Hedderich R., Ingram-Smith C., Kuettner H.C., Krzycki J.A., Leigh J.A., Li W., Liu J., Mukhopadhyay B., Reeve J.N., Smith K., Springer T.A., Umayam L.A., White O., White R.H., Conway de Macario E., Ferry J.G., Jarrell K.F., Jing H., Macario A.J., Paulsen I., Pritchett M., Sowers K.R., Swanson R.V., Zinder S.H., Lander E., Metcalf W.W., and Birren B. (2002) The genome of M. acetivorans reveals extensive metabolic and physiological diversity. Genome Res. 12 (4): 532-542.
Gelfand M.S., Koonin E.V., Mironov A.A. (2000) Prediction of transcription regulatory sites in Archaea by a comparative genomic approach. Nucleic Acids Res. 28. 695-705.
Gibson T.J., Higgins D.G., Plewniak F., Thompson J.D. and Jeanmougin F.. (1997) The Clustal_X windows interfaca: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Resesrch, Vol. 25, No. 24.
Jiang, P., Zucker P., Atkinson M. R., Kamberov E.S., Tirasophon W., Chandran P., Schefke B.R., and Ninfa A.J.. (1997). Structure/function analysis of the PII signal transduction protein of Escherichia coli: genetic separation of interactions with protein receptors. J. Bacteriol. 179:4342-4353
Kessler P.S., McLarnan Jennifer, and Leigh J.A.. (1996) Nirtogenase phylogeny and the molybdenum dependence of nitrogen fixation in Methanococcus maripaludis. Journal of Bacteriology, Vol. 179, No. 2: 541-543
Kessler P.S., Blank C., and Leigh J.A.. (1998) The nif gene operon of the Methanogenic Archaeon Methanococcus maripaludis. Journal of Bacteriology, Vol. 180, No. 6: 1504-1511
Kessler P.S. and Leigh J.A.. (1999) Genetics of nitrogen regulation in methanococcus maripaludis. Genetics 152: 1343-1351
Kessler P.S., Daniel C., and Leigh J.A.. (2000) Ammonia switch-off of nitrogen fixation in the methanogenic archaeon Methanococcus maripaludis: mechanistic features and tequirement for the novel GlnB homologues, NifI1 and NifI2.Department of Microbiology, University of Washington, Seattle, Washington 98195 Received 10 July 2000/Accepted 7 November 2000
Lie T.J., Leigh J.A.( 2002) A novel repressor of nif and glnA expression in the methanogenic archaeon Methanococcus maripaludis. J Bacteriol. 184. 5301-5306
Mironov A.A., Koonin E.V., Roytberg M.A., Gelfand M.S. (1999) Computer analysis of transcription regulatory patterns in completely sequenced bacterial genomes. Nucleic Acids Res. 27. 2981-2989.
Mironov A.A., Vinokurova N.P., Gelfand M.S. (2000) Software for analyzing bacterial genoms. Mol Biol (Mosk). 34. 253-262
Slesarev A.I., Mezhevaya K.V., Makarova K.S., Polushin N.N., Shcherbinina O.V., Shakhova V.V., Belova G.I., Aravind L., Natale D.A., Rogozin I.B., Tatusov R.L., Wolf Y.I., Stetter K.O., Malykh A.G., Koonin E.V., and Kozyavkin S.A. (2002) The complete genome of hyperthermophile Methanopyrus kandleri AV19 and monophyly of archaeal methanogens. Proc Natl Acad Sci U S A. 99 (7): 4644-4649.
Smith D.R., Doucette-Stamm L.A., Deloughery C., Lee H., Dubois J., Aldredge T., Bashirzadeh R., Blakely D., Cook R., Gilbert K., Harrison D., Hoang L., Keagle P., Lumm W., Pothier B., Qiu D., Spadafora R., Vicaire R., Wang Y., Wierzbowski J., Gibson R., Jiwani N., Caruso A., Bush D., and Reeve J.N. (1997) Complete genome sequence of Methanobacterium thermoautotrophicum deltaH: functional analysis and comparative genomics. J Bacteriol. 179 (22): 7135-7155.
